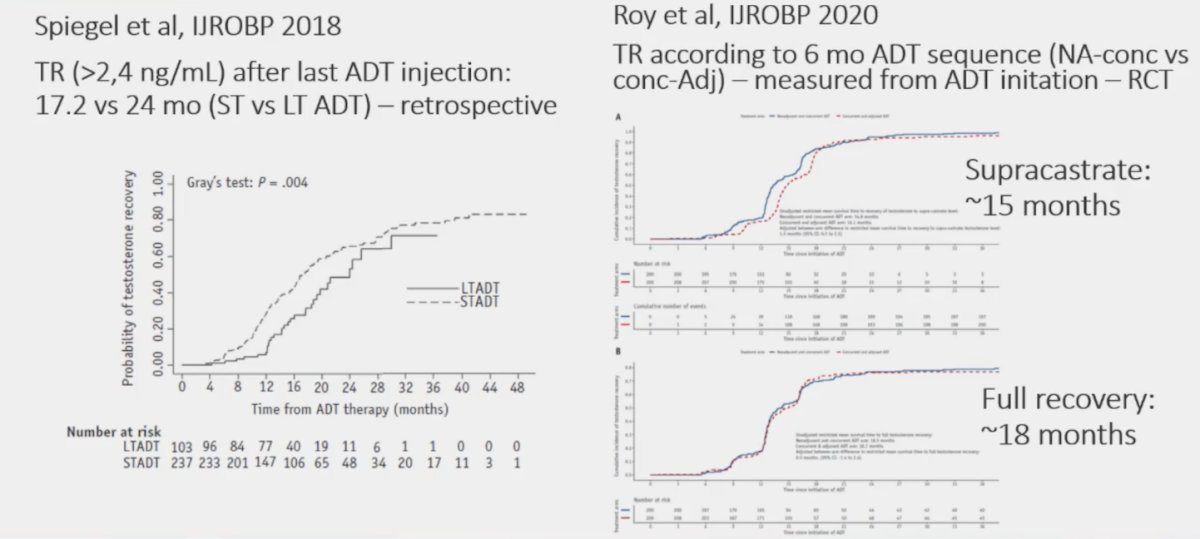
Imágenes ExA-SPIM de neuronas en una pieza de cerebro de macaco que mide 1 x 1 x 1,5 cm.Crédito: Instituto Allen de Neurodinámica
El cerebro de los mamíferos es un sistema multidominio. Los circuitos neuronales forman una supercarretera hacia la información, con algunas proyecciones que potencialmente se extienden decenas de centímetros hacia el cerebro. Pero estas proyecciones también tienen unos cientos de nanómetros de espesor, aproximadamente una milésima parte del ancho de un cabello humano.
Comprender cómo el cerebro codifica y transmite señales requiere alinear eventos en ambas escalas. Tradicionalmente, los investigadores del cerebro han abordado esto mediante un proceso de varios pasos: cortar el tejido en secciones delgadas, obtener imágenes de cada sección en alta resolución, volver a cortar las capas y reconstruir las trayectorias de las neuronas individuales.
Zhuhao Wu, neurocientífico de Weill Cornell Medicine en la ciudad de Nueva York, describe este último paso como «como rastrear un cable telefónico por Manhattan». En realidad, es «mucho más complicado», agrega, «porque cada neurona hace miles, si no decenas de miles, de conexiones».
Los fabricantes de los microscopios buscaron permitir a los investigadores echar un vistazo de gran angular a una gran sección de tejido y aún así ver los detalles de cerca, sin tener que cortar primero el tejido y luego reconstruir los axones en diferentes secciones. El desafío es que los objetivos de los microscopios generalmente están diseñados de tal manera que es difícil tomar imágenes de alta resolución de muestras grandes.
La versión inicial publicada en junio ofrece una solución1. Primero, los investigadores eliminaron químicamente la grasa para hacer que el tejido fuera translúcido. Luego lo incorporaron a un material llamado hidrogel, que absorbe agua para expandir el tejido hasta tres veces su tamaño original. Finalmente, lo escanearon con una lente prestada de un campo científico completamente diferente. De esta manera, fue posible obtener imágenes de cerebros completos de ratones sin necesidad de disecciones y con una resolución de aproximadamente 300 nm en el plano de imagen y 800 nm axialmente (perpendicular al plano), que es similar a la microscopía confocal. Una técnica ampliamente utilizada para obtener imágenes cerebrales de alta resolución. Apodado ExA-SPIM (Microscopía de iluminación plana asistida por expansión selectiva), el protocolo también se ha utilizado para obtener imágenes de neuronas en los cerebros de macacos y humanos.
“Lo mejor que trae un sistema así es la combinación de poder filmar volúmenes muy grandes [of brain tissue] «Con una resolución muy alta», dice Jayaram Chandrashekar, neurocientífico del Instituto Allen de Neurodinámica en Seattle, Washington, quien codirigió el estudio con dos colegas: el desarrollador de microscopios Adam Glaser y el presidente del instituto Karel Svoboda.
Este enfoque puede generar imágenes del cerebro de un ratón completo en menos de un día, mucho más rápido y con una resolución más alta de lo que es posible con otros métodos de cerebro completo que se han aplicado a proyecciones axonales, como MouseLight y fMost, que usan tomografía, dice Chandrashekar. . El hecho de que las imágenes requieran solo una reconstrucción computacional limitada aumenta en gran medida la precisión de los datos resultantes.
Ninguno de los componentes del sistema es nuevo, señala Wu, solo se han ensamblado de manera sinérgica. «No es el primer intento de hacer esto, pero es probablemente el mejor intento que estamos haciendo en este momento», dice.
Paso a paso
El primer elemento del protocolo, la extracción y expansión de tejido, se ha utilizado durante décadas, pero generalmente en piezas de tejido más pequeñas. El equipo tuvo que mejorar la técnica para garantizar que las muestras de cerebro se expandieran isotrópicamente, es decir, en la misma cantidad en todas las direcciones, dice Glaser.
El núcleo del nuevo método, dice Glaser, es la lente. Al elegirlo, observaron las industrias de métricas y visión artificial, y se decidieron por uno que se usa normalmente para identificar defectos del tamaño de un píxel en pantallas planas y otros dispositivos electrónicos a medida que se mueven a lo largo de una cinta transportadora. La lente tiene un campo de visión más grande que el que normalmente se usa en las ciencias de la vida, dice, y debido a que el tejido se estira antes de obtener la imagen, una resolución de un micrómetro es suficiente para rastrear un axón en todo el cerebro.
Los investigadores construyeron esta lente en un microscopio que visualizaba las estructuras 3D como una serie de paneles 2D, una técnica llamada iluminación plana selectiva. Luego agregaron una cámara, también proveniente de las industrias de visión y metrología, que tiene 38 veces más píxeles que las cámaras que se usan tradicionalmente en las ciencias de la vida y puede capturar un campo de visión que mide 10,6 mm x 8,0 mm. Con esas especificaciones, una lámina bidimensional de un cerebro de ratón ampliado se puede capturar en unos 15 cortes, en comparación con los 400 cortes de un microscopio convencional, dice Chandrashekar.
Un potente microscopio captura proteínas motoras con un detalle sin precedentes
“Me gustó que pensaran fuera de la caja y miraran otras áreas de la ciencia”, dice Flavie Lavoie-Cardenal, neurocientífica y microcientífica de la Universidad Laval en la ciudad de Quebec, Canadá. «Este es un método mejor que los métodos que involucran lentes dedicados», agrega, lo que hace que el sistema sea menos accesible para otros investigadores.
ExA-SPIM es «definitivamente importante», dice Catherine Willig, microcientífica de la Universidad Georg August en Göttingen, Alemania, particularmente para estudios de conectividad cerebral o «conexiones neuronales». Pero agrega: «Sería bueno mejorar un poco la resolución para poder ver claramente las espinas dendríticas».
enterrar los datos
Según Glaser, se pueden obtener imágenes de un cerebro en menos de un día. Hasta ahora, el equipo ha obtenido imágenes de unos 25 cerebros de ratón, lo que ha producido unos 2,5 petabytes de datos, que el equipo comprime cinco veces y almacena en la nube. Para analizar los datos, los investigadores colaboran con Google, que proporciona algoritmos de aprendizaje automático para procesar los datos y reconstruir imágenes de neuronas.
La cantidad de datos involucrados puede ser un gran obstáculo para los usuarios potenciales, dice Harry Shroff, microscopista del Janelia Research Campus en Ashburn, Virginia. Además del desafío de analizar este volumen de datos, simplemente transferirlos de un microscopio a una computadora es un gran avance, dice. «Las asociaciones de laboratorios o institutos pueden invertir en personas, o en la fuerza o la infraestructura para hacerlo, pero no es poca cosa», dice.
Microscopios inteligentes detectan biología transitoria
Sin embargo, el diseño del microscopio es de código abierto y las instrucciones para construirlo son Disponible en github. Pero Glaser dice que la versión actual es un prototipo que planea optimizar y documentar durante el próximo año. «Lleva tiempo ensamblarlo y no es muy fácil de construir», dice. Hasta el momento, varios laboratorios han expresado interés, pero ninguno ha intentado configurar el sistema. «Rediseñaremos y rediseñaremos el microscopio y lo documentaremos muy bien, y esperamos recibir fondos para implementar el sistema para que otros grupos puedan adoptarlo».
El seguimiento de las conexiones neuronales a largo plazo es la aplicación más obvia, dice Chandraskar, y una que los investigadores están buscando actualmente. Pero también están reelaborando los protocolos de limpieza y expansión para mantener intactas las moléculas vitales como el ARN y las proteínas, y permitir que también se localicen.
«Espero ver cómo otros problemas de la neurociencia también pueden beneficiarse de esta tecnología», dice Wu.

«Alborotador. Amante de la cerveza. Total aficionado al alcohol. Sutilmente encantador adicto a los zombis. Ninja de twitter de toda la vida».




More Stories
Restaurar la testosterona después de dejar el TDA: ¿quién corre el riesgo de no recuperarse a largo plazo?
Los científicos finalmente han descubierto signos de vida en un planeta dos veces más grande que la Tierra.
La niebla se encuentra con las montañas Salomón