El síndrome respiratorio agudo severo coronavirus 2 (SARS-CoV-2), el virus responsable de la enfermedad por coronavirus 2019 (COVID-19), continúa desafiando la actividad social y económica normal.
El aspecto más confuso de COVID-19 es su aparente imprevisibilidad. Aunque no causa ningún síntoma o desarrolla síntomas leves en la mayoría de las personas infectadas, COVID-19 puede causar una inflamación inmunitaria desorganizada, que puede provocar daños sistémicos e incluso la muerte en muchas otras personas.
Stady: Variación dependiente de HLA en las interacciones de las células T CD8 + del SARS-CoV-2 con los coronavirus humanos. Crédito de la imagen: Design_Cells / Shutterstock.com
Nuevo estudio publicado en servidor de preimpresión bioRxiv * Se analiza cómo las diferentes respuestas a la misma infección y las vacunas COVID-19 son probablemente el resultado de diferencias genéticas. Estas diferencias parecen influir en la naturaleza de la inmunidad preexistente al SARS-CoV-2 que resultó de una infección previa con otros coronavirus humanos estacionales endémicos (hCoV).
antecedentes
La reactividad cruzada entre los antígenos del SARS-CoV-2 y los de otros hCoV puede mejorar la respuesta inmune a la infección natural o la vacunación, o reducir la gravedad de la enfermedad, a través de las células T de memoria. Sin embargo, esta reacción cruzada también puede conducir a un aumento de la infección dependiente de anticuerpos (ADE) o un aumento de la producción de anticuerpos, lo que puede conducir a una enfermedad crónica.
Se sabe que cuatro hCoV endémicos incluyen HCoV-229E, -NL63, -OC43 y -HKU1. Estudios anteriores demostraron que las células T reactivas al SARS-CoV-2, especialmente las células CD4 + y CD8 +, se encontraron en individuos no infectados.
La evidencia de la inmunidad de las células T CD8 + al virus en individuos sin experiencia es más controvertida. El estudio actual sugiere que existe una gran posibilidad de que las células T de memoria de una infección previa por hCoV puedan reaccionar contra antígenos muy similares del SARS-CoV-2, lo que confiere protección contra este último virus. Sin embargo, esto depende de la presencia de proteínas específicas para reconocer células conocidas como células de antígeno leucocitario humano (HLA).
¿Qué mostró el estudio?
Los investigadores encontraron miles de antígenos peptídicos inmunogénicos del SARS-CoV-2 que se presentaron mediante complejos principales de histocompatibilidad (MHC) de clase I y II. Tanto las moléculas de MHC de clase I como de clase II consistían en aproximadamente un tercio de estos antígenos cada una, mientras que el tipo de MHC del tercio restante era desconocido. Se sabía que los alelos HLA de los complejos MHC eran aproximadamente 57% y 42%, respectivamente.
Al centrarse en los péptidos inmunes, los investigadores lograron identificar la información que faltaba. Al analizar las preferencias de HLA hacia los péptidos virales, los investigadores encontraron que entre los 9M, HLA-C * 07: 02 se observó con mayor frecuencia.
Un total de 126 péptidos de SARS-CoV-2 presentaron una gran similitud con aproximadamente 300 péptidos de HCoV. El primero fue presentado por múltiples alelos HLA, más de la mitad de los cuales eran 9 mM, cinco eran 15 mM y más de uno de cada siete alelos eran 10 mM.
El mayor número de coincidencias fue con HKU1 y OC43, lo que probablemente se deba a la similitud general que comparten con el genoma del SARS-CoV-2. Además, 42 péptidos de SARS-CoV-2 unidos a tres o más hCoV, junto con pequeños grupos, mostraron homología con una sola cepa.
Los copéptidos fueron más abundantes con ORF1ab viral y proteínas de pico para OC43 y HKU1. Estos resultados pertenecen a secuencias con un 70% o más de homología. Los resultados sugieren que existen formas potenciales de protegerse contra este virus a través de una inmunidad preexistente.
Variación de la proteína humana
Los investigadores encontraron que los péptidos del SARS-CoV-2 presentados por el HLA de clase I tenían una mayor variación con la proteína humana cuando eran inmunogénicos que no inmunes. Este no fue el caso de los péptidos de clase II introducidos.
Con este fin, los autores del presente estudio detectaron varios péptidos inmunogénicos del SARS-CoV-2 con una similitud promedio del 72% con los autoantígenos. Por lo tanto, las células T que reaccionan pasivamente con estos péptidos deben seleccionarse para prevenir enfermedades autoinmunes o ADE. Entre ellos se encuentran CCL3 y CCL3L1, ambos normalmente elevados durante una tormenta de citocinas COVID-19.
Los péptidos que mostraron homología con estas moléculas de señalización eran parte del propio repertorio de las células T, lo que potencialmente aumentaba el riesgo de enfermedad inmunológica. Más del 70% y casi el 80% de los aminoácidos se conservaron entre 10 y 9 M, respectivamente.
Estos péptidos merecen más investigación por su papel en la posible autoinmunidad después de la vacunación o la infección natural con COVID-19.
Interacción de células T CD8 +
Aproximadamente 250 péptidos del SARS-CoV-2 tienen receptores específicos de células T. Curiosamente, el 80% de los TCR reconocen el 16% (n = 40) de péptidos virales, 36 de los cuales son privados y el resto son compartidos por ambos grupos. En particular, se encontró que el gen V TRBV20-1 y el gen J TRBJ2-1 estaban sobreexpresados.
El histograma de sus interacciones mostró dos centros claros, ya que los sujetos sanos estaban conectados a TCR genéricos que reconocen copéptidos o péptidos del SARS-CoV-2 (principalmente el primero) en un solo centro. El segundo eje, que representa a los pacientes con COVID-19 en recuperación, mostró principalmente el repertorio general de TCR con péptidos específicos del SARS-CoV-2.
Varios TCR reconocieron péptidos compartidos por estos dos grupos, lo que indica la presencia de TCR generales para péptidos compartidos en individuos sin experiencia, así como en pacientes con COVID-19. Esto puede no indicar una inmunidad de células T preexistente, sino una reactividad cruzada.
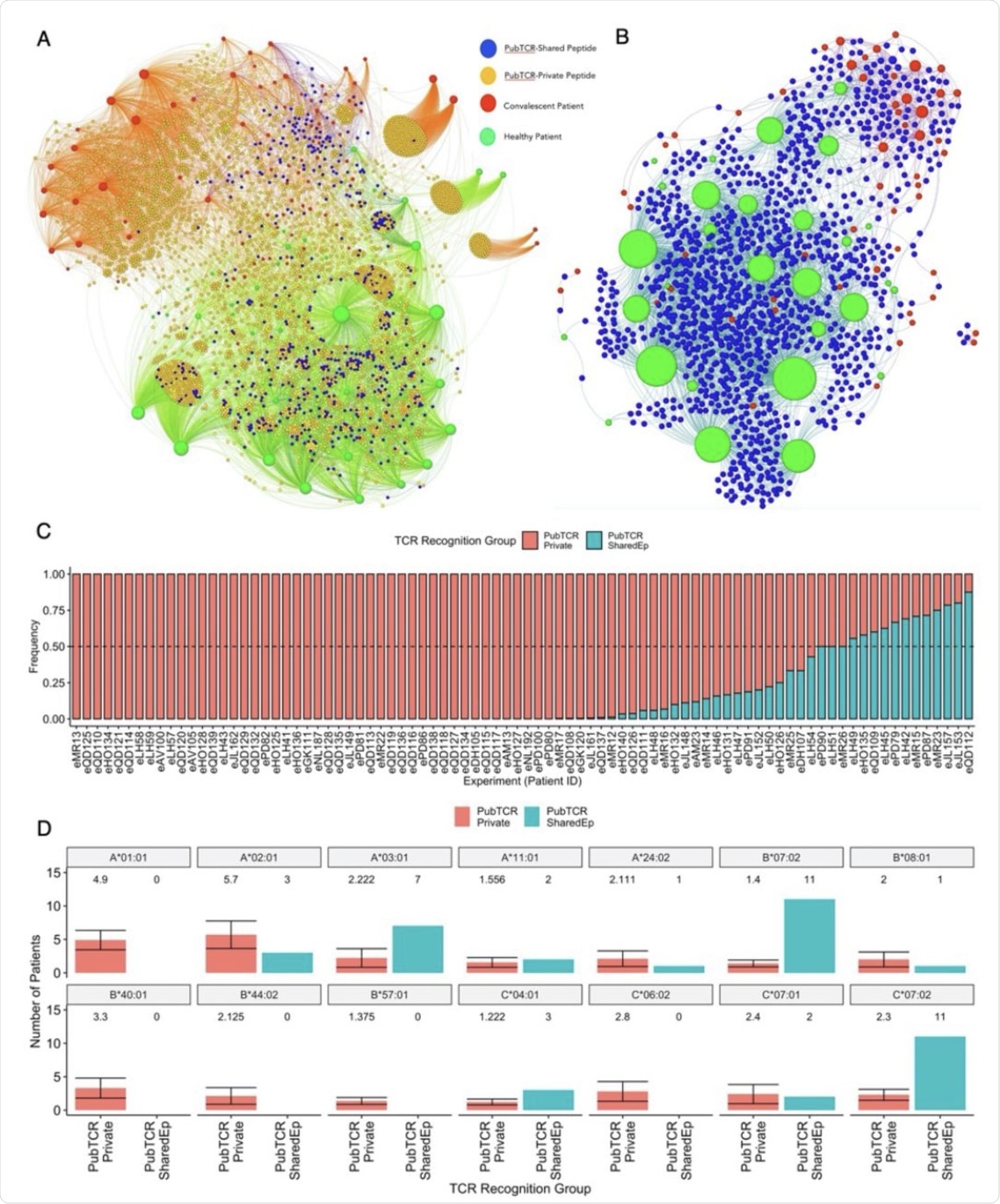
También se observa que mientras que la mayoría de los pacientes convalecientes (51/86) tenían TCR genéricos que se unían principalmente a péptidos específicos del SARS-CoV-2, 12 pacientes mostraron> 50% de TCR capaces de reconocer péptidos comunes. Estos pacientes mostraron enriquecimiento de HLA-B * 07: 02, HLA-C * 07: 02 y enriquecimiento de HLA A * 03: 01, que fue similar al grupo de alelos HLA más amplio, con una mayor representación de HLA A * 1 : 01, en el grupo de péptidos.
Juntos, estos resultados respaldan informes anteriores que muestran que la reactividad cruzada de células T CD8 + HCoV-CoV-2 puede estar condicionada por HLA. Por lo tanto, solo estos pacientes pueden tener una protección más fuerte contra el SARS-CoV-2.
Estos resultados muestran que hay un grupo de epítopos con TCR genéricos similares en controles sanos, junto con un nuevo conjunto de péptidos que provocan respuestas de células T en COVID-19. No se ve que el último grupo de péptidos se expanda en pacientes no expuestos.
Sin embargo, también hay TCR especiales en sujetos sin experiencia con homodímeros similares de SARS-CoV-2 y hCoV. Algunos de estos son de hecho TCR genéricos, reportados en estudios independientes, capaces de reactividad cruzada con homopéptidos de SARS-CoV-2 y OC43 / HKU1.
Identificación de péptidos conservados para inmunidad protectora
Los científicos también encontraron que un total de 84 péptidos eran similares a los TCR en personas críticas e ingenuas. Luego identificaron diez péptidos del SARS-CoV-2 que eran muy similares a los coronavirus similares al SARS anteriores. Es probable que estos péptidos estén presentados por múltiples alelos HLA y, por lo tanto, pueden ser responsables de la respuesta de las células T entre el 93% de la población mundial.
¿Cuáles son los efectos?
«Creemos que esto es una idea alentadora sobre la búsqueda de dianas sistémicas de células T MERS-CoV, además de sugerirlos como los principales candidatos para la inmunidad protectora transitoria.. «
Se requerirá trabajo futuro para comprender completamente cómo mejorar la inmunidad a través de respuestas reactivas de células T. También es necesario explorar si los TCR reactivos se estimularán a través de las vacunas COVID-19, ya que esto puede ayudar a los médicos a identificar a los pacientes que desarrollarán respuestas reactivas después de la vacunación.
*Nota IMPORTANTE
bioRxiv Publica informes científicos preliminares que no han sido revisados por pares y, por lo tanto, no deben considerarse concluyentes, guiar la práctica clínica / comportamiento relacionado con la salud o tratarse como información establecida.

«Alborotador. Amante de la cerveza. Total aficionado al alcohol. Sutilmente encantador adicto a los zombis. Ninja de twitter de toda la vida».





More Stories
Los médicos combinaron una bomba cardíaca y un trasplante de riñón de cerdo en una cirugía avanzada
Los proyectos científicos y aplicados en la estación espacial china son fructíferos
Un antiguo ancestro del gran tiburón blanco de 30 pies ha sido descubierto en una cantera de México